確率分布が正規分布に従うか調べたい、
二つの集団が同じ確率分布から得られたものか調べたい、
といった時に使うのが、コロモゴロフスミルノフ検定(Kolmogorov–Smirnov test)
コルモゴロフ–スミルノフ検定(コルモゴロフ–スミルノフけんてい、英: Kolmogorov–Smirnov test)は統計学における仮説検定の一種であり、有限個の標本に基づいて、二つの母集団の確率分布が異なるものであるかどうか、あるいは母集団の確率分布が帰無仮説で提示された分布と異なっているかどうかを調べるために用いられる。しばしばKS検定と略される。
1標本KS検定は、経験分布を帰無仮説において示された累積分布関数と比較する。主な応用は、正規分布および一様分布に関する適合度検定である。正規分布に関する検定については、リリフォースによる若干の改良が知られている(リリフォース検定)。正規分布の場合、一般にはリリフォース検定よりもシャピロ-ウィルク検定やアンダーソン-ダーリング検定の方がより強力な手法である。
2標本KS検定は、二つの標本を比較する最も有効かつ一般的なノンパラメトリック手法の一つである。これは、この手法が二つの標本に関する経験分布の位置および形状の双方に依存するためである。
wikipedia:コルモゴロフ-スミルノフ検定
pythonではscipyで用意されている。
1標本の場合
from scipy import stats stats.kstest(rvs, cdf, args=(), N=20, alternative='two-sided', mode='approx')
rvs:データor単語
cdf:累積分布
args:分布の形状を指すパラメータ(必要に応じて)
p値はpvalueで取り出す。
試してみる

- 正規分布
- コーシー分布
- t分布(自由度3)
の3つに対して、正確に分けられるかチェック。
正規分布のデータは正規分布だと、そうでないデータは正規分布ではないと判別してほしい。
x = stats.norm.rvs(size=500) y = stats.cauchy.rvs(size=500) z = stats.t.rvs(3,size=500) print(stats.kstest(x,"norm").pvalue) print(stats.kstest(y,"norm").pvalue) print(stats.kstest(z,"norm").pvalue)
0.611401246262 #正規分布(>0.05) 8.59754489824e-11 #コーシー分布(<0.05) 0.0135550060406 #t分布(<0.05)
正規分布とそうでない分布がきちんと判別できていることがわかる。
※帰無仮説の本来の意味からもわかるように、p値が基準値を下回らなくても正規分布と断定することはできないことに注意。
【補足】KS検定 vs. シャピロ-ウィルク検定
正規性の検定に限っては、wikipediaに載っているようにシャピロウィルク検定が良いらしい。
幸いscipyで実装されているようだったので、試してみる。
>>> from scipy import stats >>> np.random.seed(12345678) >>> x = stats.norm.rvs(loc=5, scale=3, size=100) >>> stats.shapiro(x) (0.9772805571556091, 0.08144091814756393)
- 方法
性能を比較するため、サンプルサイズを25~500の間を25刻みで増やしていく。
コーシー分布、t分布(自由度3)、正規分布のそれぞれの分布・サンプルサイズに対して40回のサンプリングを行い、得られたp値の平均値をプロットする。
どれくらいのサンプルサイズだと誤判別が起きないかを確認してみる。有意水準は95%とした。
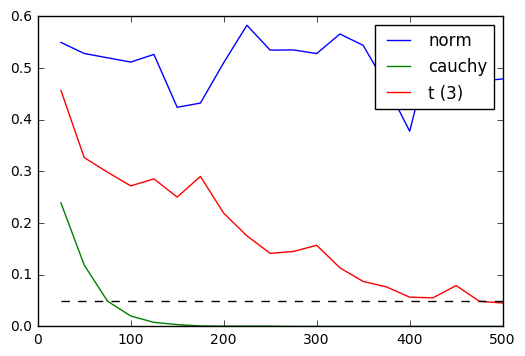
1. コロモゴロフスミルノフ検定
2. シャピロウィルク検定

コーシー分布・t分布のどちらともが点線(5%)を下回っていれば判別できている(正規分布ではないと判断している)ということだが、シャピロウィルク検定はサンプルサイズが小さくてもよく判別できているといえる。
シャピロウィルク検定の場合サンプルサイズが100程度あれば十分かも。
また、どちらの検定でも正規分布なのに正規分布でないと判断してしまうことはなかった。
使ったコード
samplesize = range(25,525,25) samplingnum = 40 xlist = [] ylist = [] zlist = [] for i in samplesize: xsampling = [] ysampling = [] zsampling = [] for j in range(samplingnum): x = stats.norm.rvs(size=i) y = stats.cauchy.rvs(size=i) z = stats.t.rvs(3, size=i) xsampling.append(stats.shapiro(x)[1]) ysampling.append(stats.shapiro(y)[1]) zsampling.append(stats.shapiro(z)[1]) xlist.append(sum(xsampling)*1.0/samplingnum) ylist.append(sum(ysampling)*1.0/samplingnum) zlist.append(sum(zsampling)*1.0/samplingnum) plt.plot(samplesize,xlist, label="norm") plt.plot(samplesize,ylist, label="cauchy") plt.plot(samplesize,zlist, label="t (3)") plt.plot(samplesize,[0.05 for i in range(len(samplesize))], "--", color="black") plt.legend() plt.show()
2標本の比較
from scipy import stats stats.ks_2samp(data1, data2)
データサイズは違っても構わない。
試してみる
①コーシー分布とコーシー分布
②コーシー分布とt分布(自由度3)
で比べてみる
y = stats.cauchy.rvs(size=500) y1 = stats.cauchy.rvs(size=500) z = stats.t.rvs(3, size=500) print(stats.ks_2samp(y,y1).pvalue) print(stats.ks_2samp(y,z).pvalue)
0.655550311167 # コーシー分布vsコーシー分布(>0.05) 0.022458470313 # コーシー分布vst分布(<0.05)
5%の基準だと、きちんと判別できている。
公式サイト:
scipy.stats.shapiro — SciPy v0.19.0 Reference Guide
scipy.stats.kstest — SciPy v0.14.0 Reference Guide
scipy.stats.ks_2samp — SciPy v0.15.1 Reference Guide